近期,西北民族大学生物医学研究中心食品生物技术团队在国际学术期刊Food Bioscience(农林科学2区TOP期刊)发表了题为“Microbial dynamics and metabolic changes during Qula fermentation from skimmed yak milk”的研究论文,该研究建立了曲拉发酵过程中微生物组成与代谢产物之间的相关性,揭示了微生物群落和代谢通路对曲拉风味形成的重要作用。
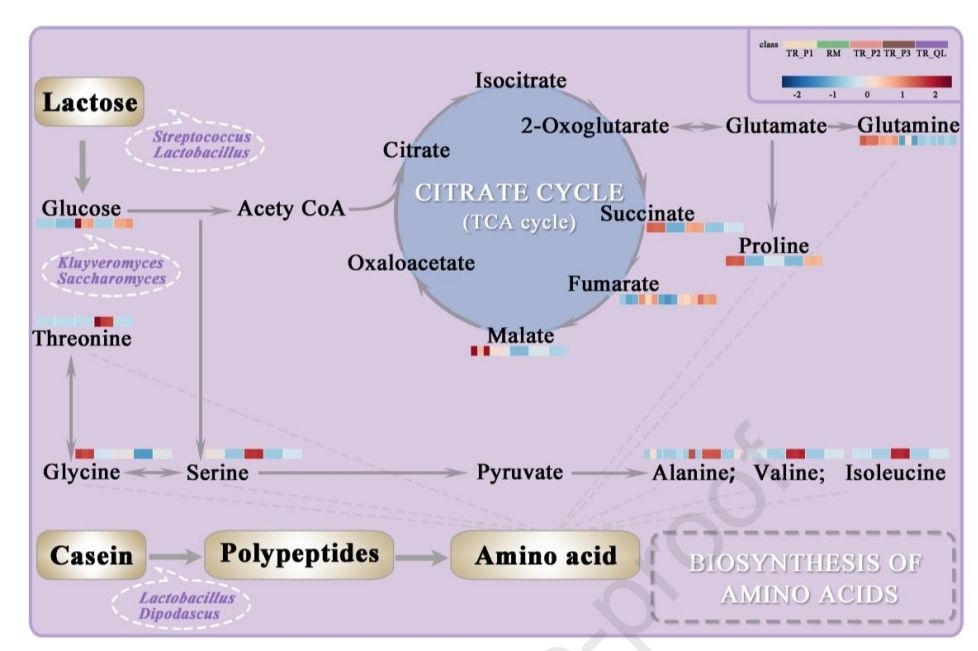
研究运用16S rRNA 基因的高通量测序和非靶向代谢组学建立了曲拉发酵过程中微生物组成与代谢产物之间的相关性。结果显示,未发酵的牦牛乳(RM)表现出最高的细菌多样性,而发酵 8 小时(TR_P2)的样品中细菌相对丰度最低;干曲拉(TR_QL)中真菌多样性最高。代谢组学分析鉴定出 65 种差异丰度代谢物 (p 值<0.05,VIP≥1)贯穿5个发酵阶段,包括有机酸、类脂分子、有机氧化合物等。“氨基酸的生物合成”和“缬氨酸、亮氨酸和异亮氨酸的生物合成”途径与 TR_P2 密切相关,是曲拉品质形成的关键时期。微生物组成与代谢的相关性表明微生物群分属链球菌属、乳杆菌属、克鲁维酵母属、酵母菌属和双足囊菌属5个核心属。本研究首次全面探究了曲拉发酵过程中的真菌动力学特征,以及生产过程中相关代谢产物与细菌菌群之间的关系来阐明不同微生物对曲拉风味的影响。
生物医学研究中心食品生物技术团队刘红娜教授为论文通讯作者,硕士研究生梁晓琳为论文第一作者。该研究得到中央高校基本科研业务费专项资金(31920240098)的支持。
文章链接:https://doi.org/10.1016/j.fbio.2025.107141
